Erhard Dinhobl, Stefan Reiterer
Beschreibung
Mithilfe der im VisLab erstellten Software soll es möglich sein, CT-Daten zu visualisieren. Im Folgenden ist nun die Funktionsweise und Bedienung unserer Applikation etwas genauer beschieben.
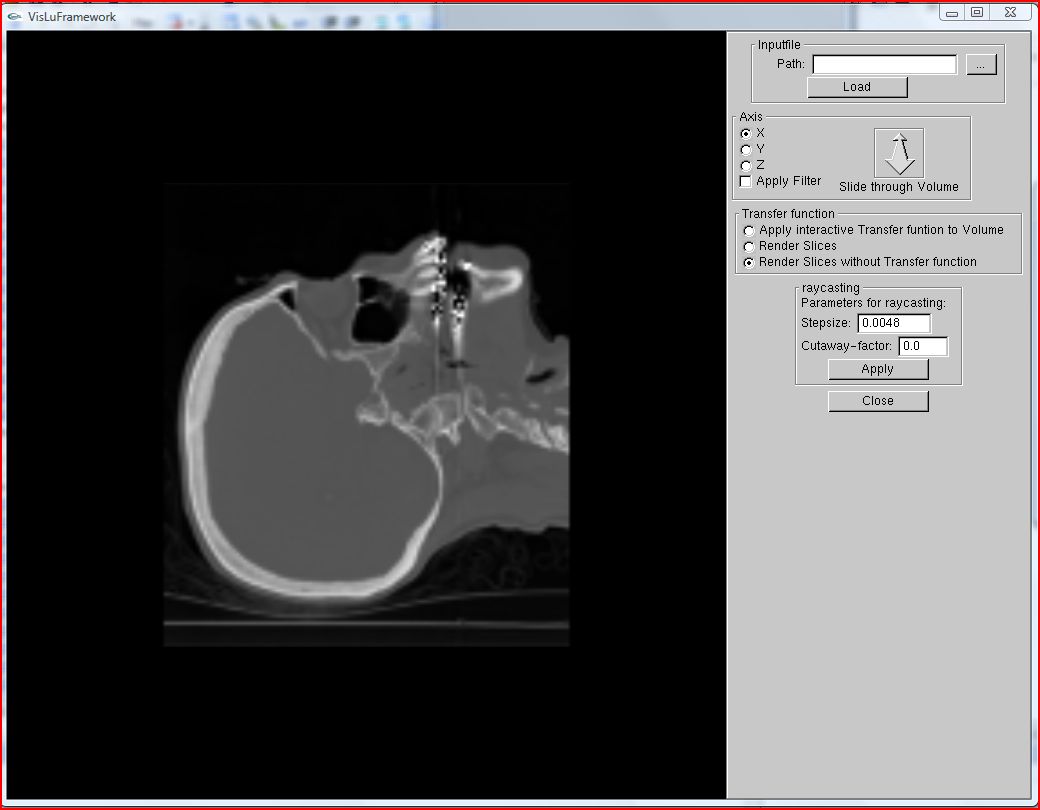
Hauptinterface

Dies ist das eigentliche Interface der Software. Im Frame "Inputfile" kann man verschiedene Datensätze laden. Weiters ist es möglich den Pfad einer Datendatei direkt in das Eingabefenster einzufügen. Wenn der Benutzer auf den Button 'Load' drückt, wird der Datensatz geladen. Dies kann einige Sekunden in Anspruch nehmen. Der Grund dafür ist, dass nun folgende Aufgaben im Hintergrund erledigt werden:
Die Optionen im Frame "Axis" dienen dazu, um im "Render Slices" Modus durch die einzelnen Schichten der 3D-Textur zu navigieren. Auf die Slices wird dabei unsere Transferfunktion angewendet. Jenachdem welche Achse gewählt wurde, wird der Datensatz von verschiedenen Seiten betrachtet. "Apply filter" ist lediglich dazu da, um die Anzeige zu glätten. Wenn der Bentzer diese Checkbox aktiviert, wird ein Gaußfilter auf die Slices angewendet.
Der Modus "Render Slices without Transfer function" rendert die Slices wie im "Render Slices" Modus und fügt keine Farbinformationen, die im Histogramm angegeben wurden hinzu. Sprich hier wird keine Transferfunktion angewendet.
Im "Apply interactive Transfer function to Volume" Modus wird nun der Datensatz in 3D angezeigt. Hierfür wird unsere Scene drei mal gerendert. In den einzelnen Renderdurchläufen werden dabei folgende Aufgaben ausgeführt.
Für das Raycasting können bei uns folgende Parameter über das User-Interface eingestellt werden.
Der Parameter "stepsize" gibt an, in welche Schritten, das Raycasring stattfinden soll. Der "Cutaway-factor" gibt an, ab welcher 'Tiefe' der Strahl gestartet wird. Dadurch kann man einfache cCutaway-Ansichten erstellen.
Das Histogramm

Mittels des Histogramms können wir interaktv eine Transferfunktion erstellen. Hierbei werden im Histogramm an der X-Achse die Dichtewerte (von 0 - 4095) angezeigt. Für bestimmte Dichtewerte kann an der Y-Achse die Transparenz gesetzt werden. Weiters kann für jeden Punkt, eine Farbe gesetzt werden. Die Farben und Transparenzwerte zwischen zwei Punkten werden linear Interpoliert. Unterhalb des Histogramms befinden sich zwei Balken. Der untere Balken stellt die Dichtewerte dar. Schwarz stellt hier Gewebe mit geringer Dichte (Blutgefäße, Haut) und weis Gewebe mit hoher Dichte (Zähne, Knochen) dar. Die Farbe kann mittels Color-Chooser gewählt werden.
Beispielbilder
Auf diesem Bild kann man die Cutaway-Ansicht gut erkennen. Das linke Bild wurde mit einem Cutaway-Faktor von 30 gerendert. Das rechte mit einem Cutaway-Faktor von 40. Hier kann man gut erkennen, dass bei einem großen Faktor mehr weggeclipt wird, als bei einem großen.


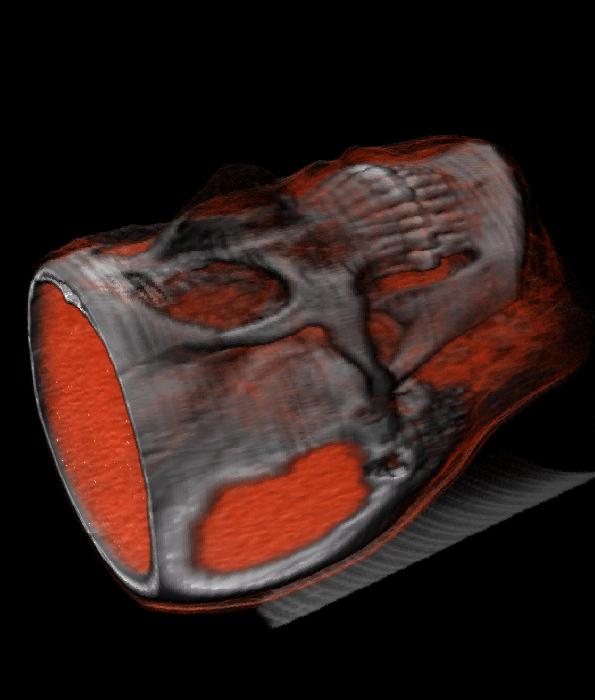
Hier sind noch ein einige Beispielbilder, die wir mit unserer Applikation erstellt haben.
Datensatz: skewed_head
.jpg)
.jpg)
.jpg)
Datensatz: stagbeetle277x277x164.dat
.jpg)
.jpg)
.jpg)
.jpg)
Download
Wer nun Lust bekommen hat, die Software selber auszuprobieren, kann sie hier herunterladen.